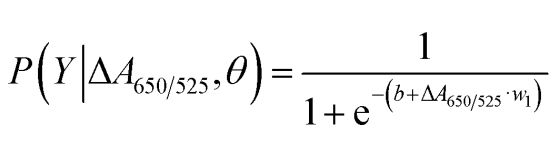Tidlig deteksjon av kreft basert på flytende biopsi er en ny retning innen kreftdeteksjon og -diagnose som er foreslått av US National Cancer Institute de siste årene, med mål om å oppdage tidlig kreft eller til og med precancerøse lesjoner. Det har blitt mye brukt som en ny biomarkør for tidlig diagnose av ulike maligniteter, inkludert lungekreft, mage-tarmtumorer, gliomer og gynekologiske svulster.
Fremveksten av plattformer for å identifisere metyleringslandskapsbiomarkører (Methylscape) har potensial til å forbedre eksisterende tidlig screening for kreft betydelig, og plassere pasienter i det tidligst behandlingsbare stadiet.
Nylig har forskere utviklet en enkel og direkte sensorplattform for deteksjon av metyleringslandskap basert på cysteamin-dekorerte gullnanopartikler (Cyst/AuNP-er) kombinert med en smarttelefonbasert biosensor som muliggjør rask tidlig screening av et bredt spekter av svulster. Tidlig screening for leukemi kan utføres innen 15 minutter etter DNA-ekstraksjon fra en blodprøve, med en nøyaktighet på 90,0 %. Artikkeltittelen er Rask deteksjon av kreft-DNA i menneskelig blod ved bruk av cysteamin-kappede AuNP-er og en maskinlæringsaktivert smarttelefon.
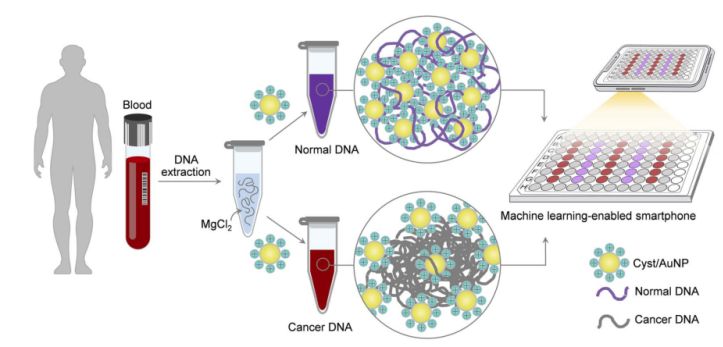
Figur 1. En enkel og rask sensorplattform for kreftscreening via cyste-/aunps-komponenter kan oppnås i to enkle trinn.
Dette er vist i figur 1. Først ble en vandig løsning brukt til å løse opp DNA-fragmentene. Cyst/AuNP-er ble deretter tilsatt den blandede løsningen. Normalt og ondartet DNA har forskjellige metyleringsegenskaper, noe som resulterer i DNA-fragmenter med forskjellige selvorganiseringsmønstre. Normalt DNA aggregerer løst og aggregerer til slutt Cyst/AuNP-er, noe som resulterer i den rødforskjøvede naturen til Cyst/AuNP-er, slik at en fargeendring fra rød til lilla kan observeres med det blotte øye. I motsetning til dette fører den unike metyleringsprofilen til kreft-DNA til produksjon av større klynger av DNA-fragmenter.
Bilder av 96-brønnsplater ble tatt med et smarttelefonkamera. Kreft-DNA ble målt av en smarttelefon utstyrt med maskinlæring sammenlignet med spektroskopibaserte metoder.
Kreftscreening i ekte blodprøver
For å utvide nytten av sensorplattformen, brukte forskerne en sensor som klarte å skille mellom normalt og kreftfremkallende DNA i ekte blodprøver. Metyleringsmønstre på CpG-steder regulerer epigenetisk genuttrykk. I nesten alle krefttyper er det observert at endringer i DNA-metylering og dermed i uttrykket av gener som fremmer tumorigenese veksler.
Som en modell for andre kreftformer assosiert med DNA-metylering, brukte forskerne blodprøver fra leukemipasienter og friske kontrollpersoner for å undersøke effektiviteten til metyleringslandskapet i å differensiere leukemiske kreftformer. Denne metyleringslandskapsbiomarkøren overgår ikke bare eksisterende raske leukemiscreeningmetoder, men demonstrerer også muligheten for å utvide til tidlig deteksjon av et bredt spekter av kreftformer ved hjelp av denne enkle og greie analysen.
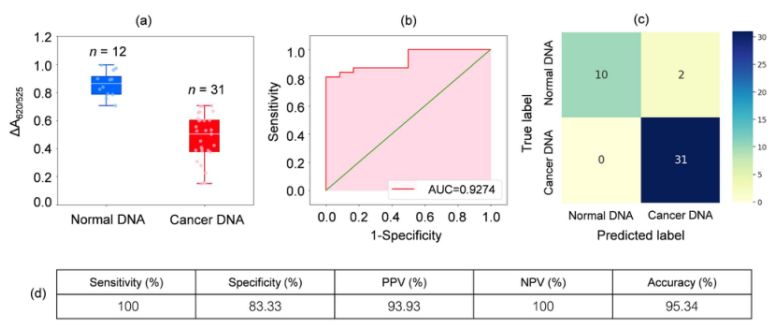
DNA fra blodprøver fra 31 leukemipasienter og 12 friske individer ble analysert. Som vist i boksplottet i figur 2a, var den relative absorbansen til kreftprøvene (ΔA650/525) lavere enn for DNA fra normale prøver. Dette skyldtes hovedsakelig den økte hydrofobisiteten som førte til tett aggregering av kreft-DNA, noe som forhindret aggregering av cyster/AuNP-er. Som et resultat ble disse nanopartiklene fullstendig dispergert i de ytre lagene av kreftaggregatene, noe som resulterte i en ulik dispersjon av cyster/AuNP-er adsorbert på normale og kreft-DNA-aggregater. ROC-kurver ble deretter generert ved å variere terskelen fra en minimumsverdi på ΔA650/525 til en maksimumsverdi.
Figur 2.(a) Relative absorbansverdier for cyste-/AuNP-løsninger som viser tilstedeværelsen av normalt (blått) og kreft-DNA (rødt) under optimaliserte forhold.
(DA650/525) av boksplott; (b) ROC-analyse og evaluering av diagnostiske tester. (c) Forvirringsmatrise for diagnostisering av normale pasienter og kreftpasienter. (d) Sensitivitet, spesifisitet, positiv prediktiv verdi (PPV), negativ prediktiv verdi (NPV) og nøyaktighet for den utviklede metoden.
Som vist i figur 2b viste arealet under ROC-kurven (AUC = 0,9274) som ble oppnådd for den utviklede sensoren høy sensitivitet og spesifisitet. Som det fremgår av boksplottet, er det laveste punktet som representerer den normale DNA-gruppen ikke godt atskilt fra det høyeste punktet som representerer kreft-DNA-gruppen. Derfor ble logistisk regresjon brukt til å skille mellom de normale gruppene og kreftgruppene. Gitt et sett med uavhengige variabler, estimerer den sannsynligheten for at en hendelse inntreffer, for eksempel en kreft- eller normalgruppe. Den avhengige variabelen varierer mellom 0 og 1. Resultatet er derfor en sannsynlighet. Vi bestemte sannsynligheten for kreftidentifikasjon (P) basert på ΔA650/525 som følger.
hvor b=5,3533,w1=-6,965. For klassifisering av utvalg indikerer en sannsynlighet på mindre enn 0,5 et normalt utvalg, mens en sannsynlighet på 0,5 eller høyere indikerer et kreftutvalg. Figur 2c viser forvirringsmatrisen generert fra «leave-it-alone»-kryssvalideringen, som ble brukt til å validere stabiliteten til klassifiseringsmetoden. Figur 2d oppsummerer den diagnostiske testevalueringen av metoden, inkludert sensitivitet, spesifisitet, positiv prediktiv verdi (PPV) og negativ prediktiv verdi (NPV).
Smarttelefonbaserte biosensorer
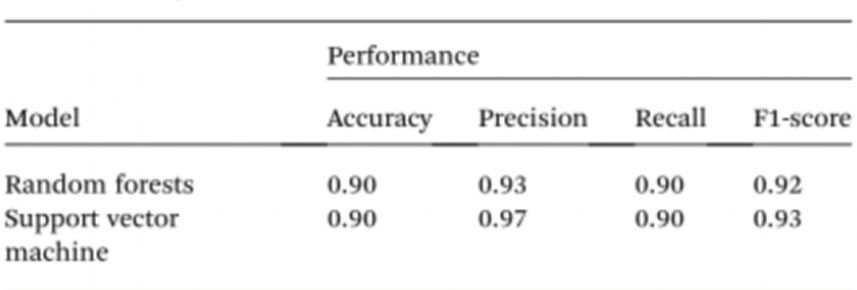
For å forenkle prøvetesting ytterligere uten bruk av spektrofotometre, brukte forskerne kunstig intelligens (KI) til å tolke fargen på løsningen og skille mellom normale og kreftrammede individer. Gitt dette ble datasyn brukt til å oversette fargen på Cyst/AuNPs-løsningen til normalt DNA (lilla) eller kreftrammet DNA (rødt) ved hjelp av bilder av 96-brønnsplater tatt gjennom et mobiltelefonkamera. Kunstig intelligens kan redusere kostnader og forbedre tilgjengeligheten ved tolkning av fargen på nanopartikkelløsninger, og uten bruk av optisk maskinvare eller smarttelefontilbehør. Til slutt ble to maskinlæringsmodeller, inkludert Random Forest (RF) og Support Vector Machine (SVM), trent til å konstruere modellene. Både RF- og SVM-modellene klassifiserte prøvene korrekt som positive og negative med en nøyaktighet på 90,0 %. Dette antyder at bruk av kunstig intelligens i mobiltelefonbasert biosensing er fullt mulig.
Figur 3. (a) Målklasse for løsningen registrert under prepareringen av prøven for bildeopptakstrinnet. (b) Eksempelbilde tatt under bildeopptakstrinnet. (c) Fargeintensiteten til cyste/AuNP-løsningen i hver brønn på 96-brønnsplaten ekstrahert fra bildet (b).
Ved hjelp av cyster/aunp-er har forskere utviklet en enkel sensorplattform for deteksjon av metyleringslandskap og en sensor som er i stand til å skille normalt DNA fra kreft-DNA når man bruker ekte blodprøver til leukemiscreening. Den utviklede sensoren viste at DNA ekstrahert fra ekte blodprøver raskt og kostnadseffektivt kunne oppdage små mengder kreft-DNA (3 nM) hos leukemipasienter på 15 minutter, og viste en nøyaktighet på 95,3 %. For å forenkle prøvetesting ytterligere ved å eliminere behovet for et spektrofotometer, ble maskinlæring brukt til å tolke fargen på løsningen og skille mellom normale og kreftpasienter ved hjelp av et mobiltelefonfotografi, og en nøyaktighet ble også oppnådd på 90,0 %.
Referanse: DOI: 10.1039/d2ra05725e
Publisert: 18. februar 2023
 中文网站
中文网站